Motivation
- 首先,由于大量的部分标记图像,我们将所有未标记的器官像素与背景标记合并,形成了边际损失。
- 其次,关于器官相互排斥的众所周知的先验知识边缘,我们设计了一个排除损失,该损失会在每个图像像素上添加排除信息,以进一步减少分割误差。
Methods
常规cross-entropy loss和常规Dice loss
输入结果通过softmax获得后验概率: \[ p_{n}=\frac{\exp \left(a_{n}\right)}{\sum_{k \in \Omega_{N}} \exp \left(a_{k}\right)}, n \in \Omega_{N} \] cross-entropy loss: \[ L_{r C E}=-\sum_{n \in \Omega_{V}} y_{n} \log \left(p_{n}\right) \] Dice loss: \[ L_{r \text { Dice }}=\sum_{n \in \Omega_{N}}\left(1-2 \cdot \frac{y_{n} p_{n}}{y_{n}+p_{n}}\right) \]
Marginal loss
通过下图方式将不同类别预测标签融合在一起。
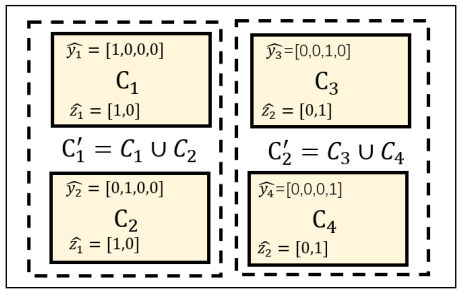
合并后的类别m的marginal概率为: \[ q_{m}=\sum_{n \in \Phi_{m}} p_{n} \] 进一步生成对应的one-hot标签
使用定义好的标签进行训练: \[ \begin{array}{l} L_{m C E}=-\sum_{m \in \Omega_{M}^{\prime}} z_{m} \log \left(q_{m}\right) \\ L_{m D i c e}=\sum_{m \in \Omega_{M}}\left(1-2 \cdot \frac{z_{m} q_{m}}{z_{m}+q_{m}}\right) \end{array} \]
问题
- 这里作者使用one-hot标签进行训练,那么是否可以考虑使用软标签进行训练?
- 为什么称之为边际损失?
Exclusion loss
在多器官分割任务中,某些类是相互排斥的。 排他性损失旨在将排他性作为每个图像像素的附加先验知识添加。
排除损失标签:
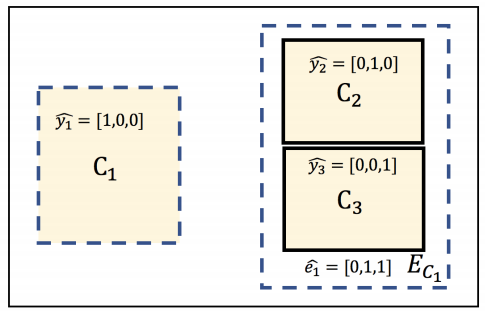
Dice loss: \[ L_{e D i c e}=\sum_{n \in \Omega_{N}} 2 \cdot \frac{e_{n} \cdot p_{n}}{e_{n}+p_{n}} \] Cross-entropy loss: \[ L_{e C E}=\sum_{n \in \Omega_{N}} e_{n} \log \left(p_{n}+\epsilon\right) \]
Experiments
数据集
数据集\(F\)表示全器官标注数据集,\(P_i\)表示单个器官标注的数据集。
数据集\(F\)
采用Multi-Atlas Labeling Beyond the Cranial Bault - Workshop and Challenge全标注数据集。包含13个器官的分割标签,肝脏、脾脏、胰腺、右肾、左肾和其他器官(胆囊、食道、胃、主动脉、下腔静脉、门静脉、脾静脉、右肾上腺、左肾上腺)。
数据集\(P_1\)
采用Decathlon-10数据集中的task03肝脏数据集。将肿瘤合并为器官作为两类(肝脏和背景)的数据集。
数据集\(P_2\)
采用task09脾脏数据集。包含41个带有脾脏分割标签的CT。
数据集\(P_3\)
采用task07胰腺数据集。见癌标签合并到胰腺获得两类(胰腺和背景)的数据集。
数据集\(P_4\)
将KiTS肾脏数据集。手动将数据分为左肾和右肾,并将癌症标签合并到相应的肾脏标签中。
所有数据集重采样为\((1.5\times 1.5 \times 3)mm^2\)。
数据集切分:\(F\)中随机抽取6个样本,\(P_1\)中随机抽取26个样本,\(P_3\)中随机抽取56个样本,\(P_4\)随机抽取42个样本作为测试,其余用于训练。
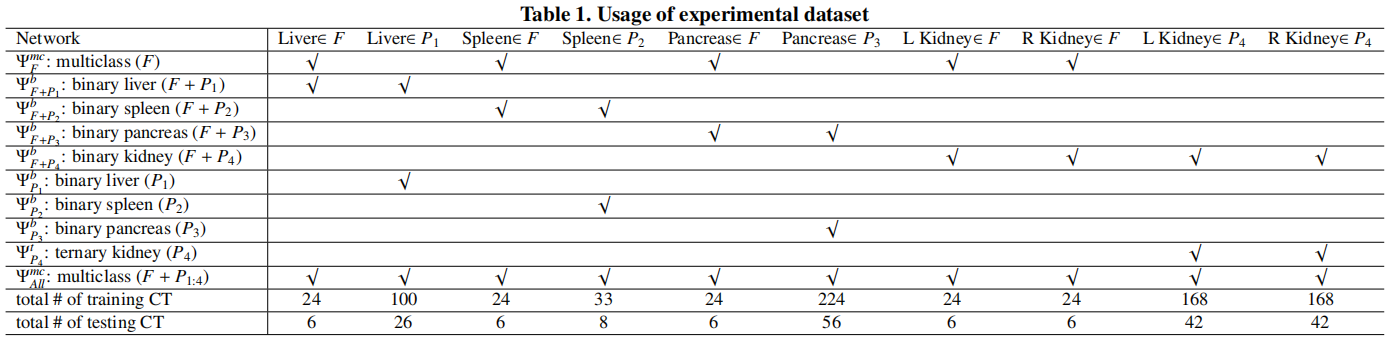
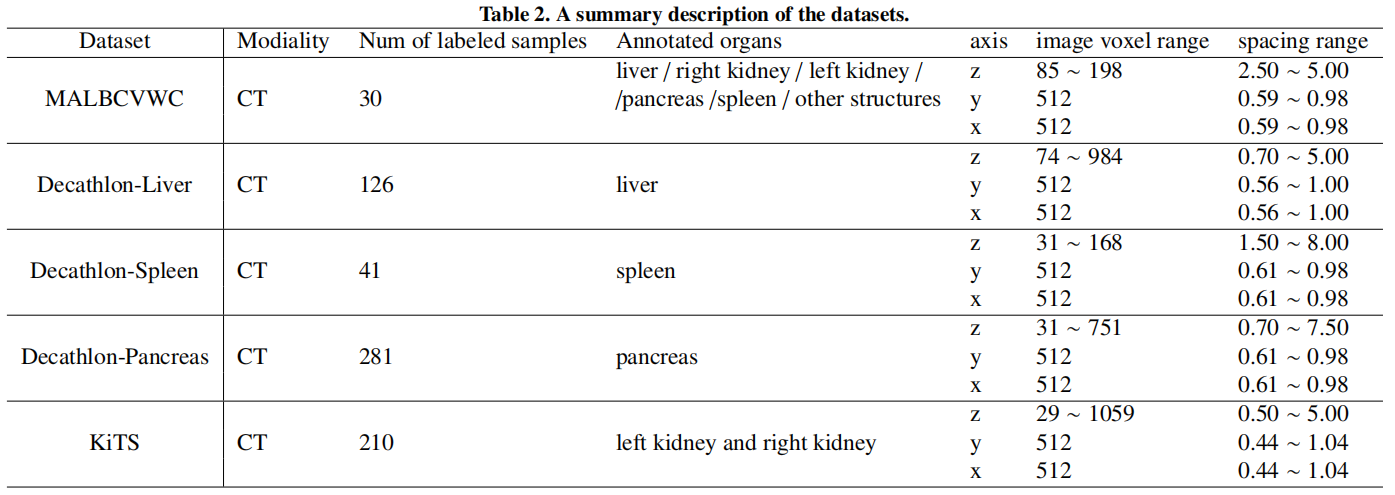
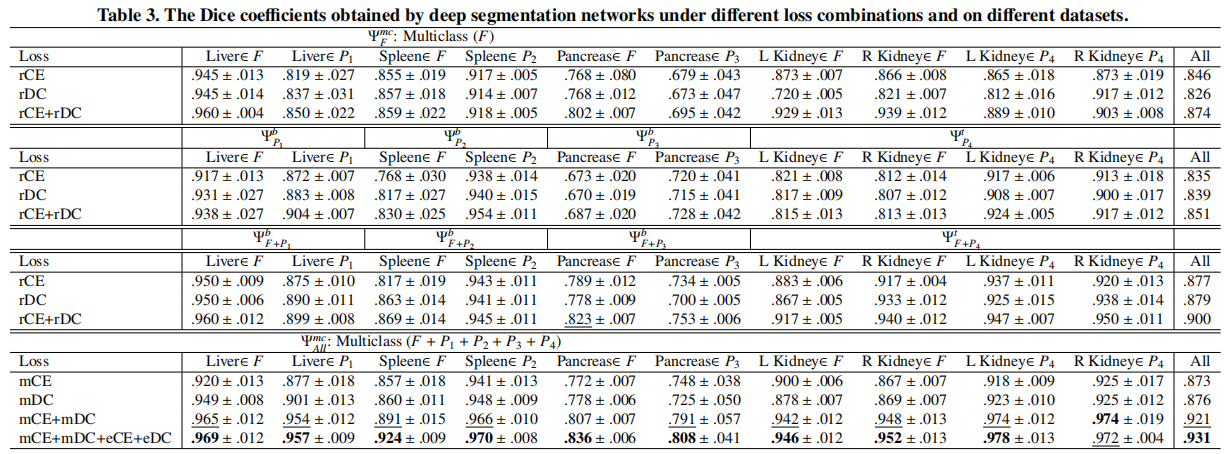
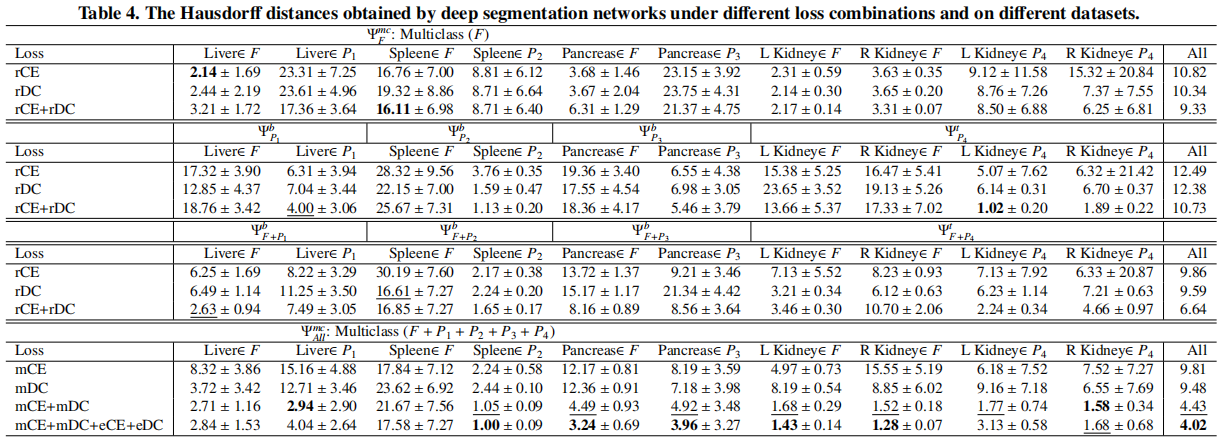
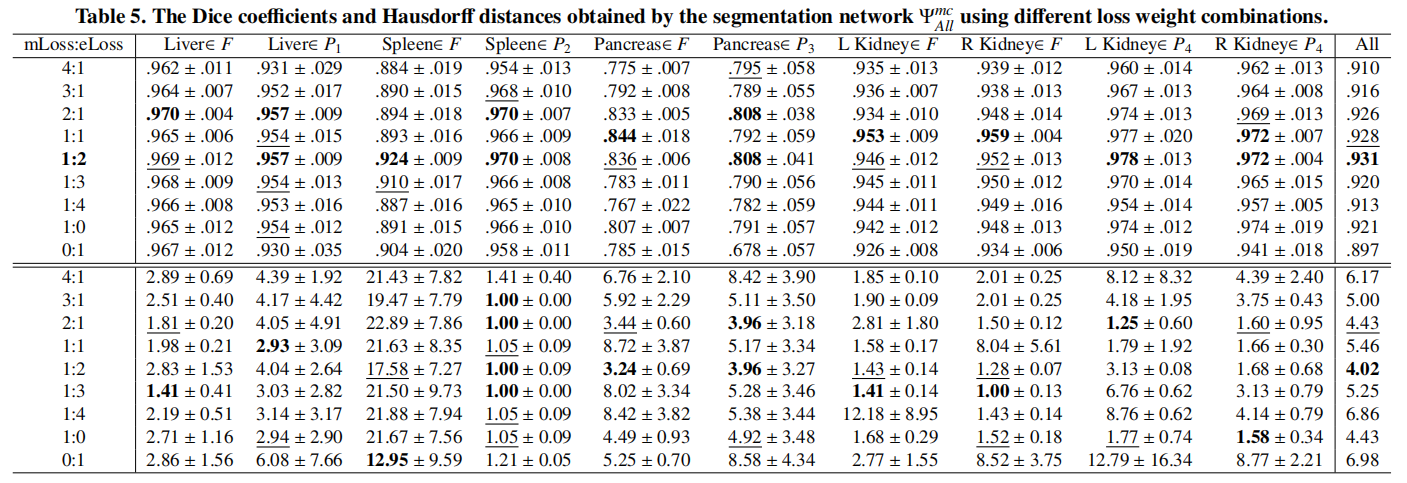
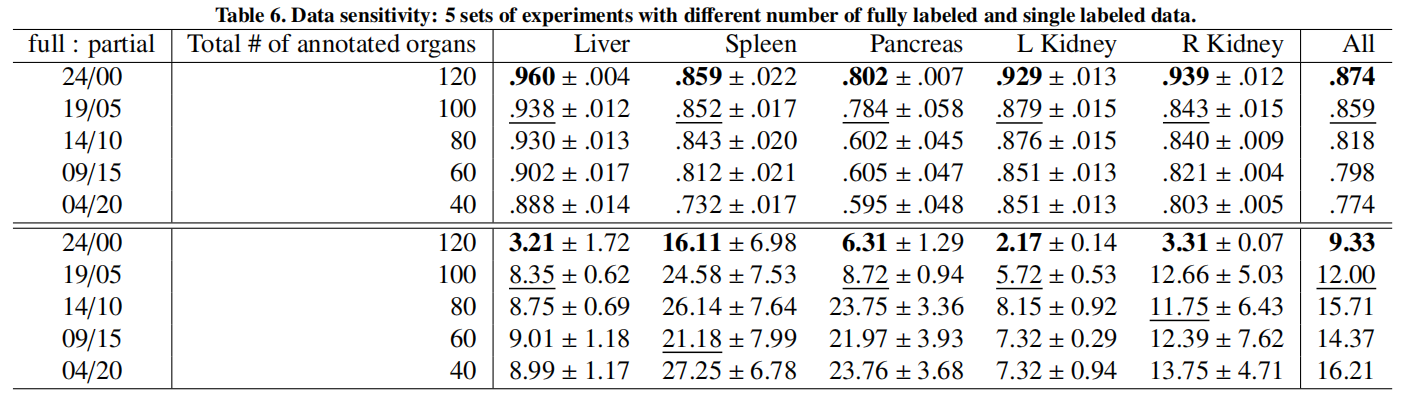
实验结果